首頁>尋醫(yī)·問藥>醫(yī)訊同期聲醫(yī)訊同期聲
3000個基因就能制造出一個完整的人類?
我們?nèi)祟惪偸菚J為,相比地球上其它活的物種而言我們處于最頂端,生命從單細胞生物開始進化了超過30億年,直到如今地球上具有多種形態(tài)尺寸以及不同能力的植物和動物,此外,隨著生態(tài)復(fù)雜性的增加,在生命的歷史長河中,我們看到了智力、復(fù)雜的社會能力以及科技創(chuàng)新的不斷發(fā)展。
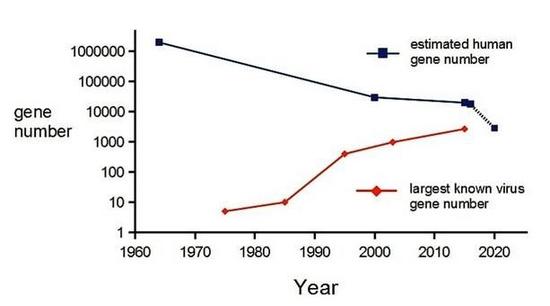
從自然角度來講,生命歷史的進展是從簡單到復(fù)雜,而且這也正反映在了基因數(shù)量的增多上,我們以卓越的智慧和全球的統(tǒng)治力為我們的發(fā)展帶路,人類是最復(fù)雜的生物,其有著一套非常精細化的基因群體。大約在半個世紀前,有研究推測人類機體中的基因有數(shù)百萬個,如今這些基因的數(shù)量下降到了2萬個,如今我們都知道,比如說香蕉都有3萬個基因,其基因數(shù)量大約是人類基因的1.5倍。隨著研究者們深入研究,他們設(shè)計出了既能夠計算有機體基因數(shù)量,又能夠發(fā)現(xiàn)不必要的基因的特殊方法。
對基因進行計數(shù)
我們可以認為機體中所有的基因都是我們烹飪的食譜,其以DNA堿基對的形式被書寫(ATCG),這些基因能夠提供提供指令來指導(dǎo)機體合成組成機體的蛋白質(zhì)并且行使著不同的功能;一個標準的基因需要大約1000個堿基字母,結(jié)合不同的環(huán)境,基因往往會負責(zé)其所扮演的不同角色,因此到底有多少基因能夠構(gòu)成一個完整的有機體呢?
當(dāng)我們談?wù)摰交驍?shù)量時,我們常常會表示我們能對病毒進行實際計數(shù),但目前研究者對真核生物進行計數(shù)面臨的一個挑戰(zhàn)就是我們機體的基因或許并不是像鴨子一樣連續(xù)地排成了一隊。我們機體烹飪書中遺傳食譜的排列方式往往會被打斷,并且會混入30萬個其它的字母,大約50%實際上都被描述為失活死亡的病毒,因此在真核生物中很難對具有關(guān)鍵功能基因進行計數(shù)。
相比較而言,對病毒和細菌基因進行計數(shù)就簡單地多,這是因為基因的原料—核苷酸對于小型生物而言非常珍貴,因此小型生物在進化過程中會通過較強的選擇剔除掉那些不必要的序列,實際上對于病毒而言最大的挑戰(zhàn)就是如何最初發(fā)現(xiàn)它們,令人驚訝的是,所有主要病毒的發(fā)現(xiàn),包括HIV等,都并不是通過測序?qū)崿F(xiàn)的,而是通過一些古老的方法,比如說將其放大到可見程度或者觀察期形態(tài)學(xué)特征,分子生物學(xué)技術(shù)的發(fā)展教會了研究者如何探索病毒世界的多樣性,但其僅僅能夠幫助我們對已知存在的病毒的基因進行計數(shù)。
越少越繁榮
在人類整個基因組中,需要維持健康生命/生活的基因數(shù)量實際上要遠遠少于當(dāng)前所估計的2萬個基因,近日就有研究進行研究發(fā)現(xiàn),維持人類生命的必要基因的數(shù)量或許非常少。研究人員對數(shù)千名個體進行研究,尋找其基因組中天然發(fā)生的“敲除”現(xiàn)象,即尋找那些“不在場”的特殊基因,我們所有的基因都有著連個拷貝,其中一個來自上一輩,通常情況下如果當(dāng)基因另一個拷貝失活時,另一個活性拷貝就會進行功能性的彌補,研究者很難發(fā)現(xiàn)攜帶兩個基因拷貝都失活的個體,因為失活的基因在天然狀態(tài)下非常罕見。
研究敲除基因的功能在實驗室對大鼠進行研究就可以,研究者們會利用現(xiàn)代的遺傳工程技術(shù)來對目的基因進行失活操作,或者移除該基因來觀察該基因失活后機體的表現(xiàn);但對人類的研究需要生活在21世紀醫(yī)療技術(shù)時代的人群,而且還需要找到適合進行遺傳學(xué)和統(tǒng)計學(xué)研究分析的譜系群體,冰島人就是一類非常有用的研究對象。
研究者通過研究發(fā)現(xiàn),有700多個基因被敲除后并不會帶來明顯的健康影響,比如,研究者指出,名為PRDM9的基因在小鼠的生育能力上扮演著重要作用,但如果在人類機體中被敲除后卻不會引發(fā)任何疾病癥狀。基于對人類敲除研究進行推測性分析,研究這表示,實際上人類僅需要3000個基因就能夠制造出一個健康完整的自己,這就類似于一種“巨大病毒”— 潘多拉病毒,2014年該病毒從3萬年的西伯利亞冰川中復(fù)蘇過來,其是目前研究者知道的最大的病毒,該病毒有2500個基因。
那么我們還需要什么基因呢?我們甚至并不知道四分之一的人類基因到底能干什么?到底其發(fā)揮著什么樣的作用?
復(fù)雜源于簡單
但是是否人類基因真正的數(shù)量時2萬?3000個?或者是別的數(shù)量呢?問題的焦點就是當(dāng)我們理解基因的復(fù)雜性時,大小或許真的并不重要了。數(shù)學(xué)家Alan Turing提出了一種多細胞發(fā)育的理論,他研究了簡單的數(shù)學(xué)模型,名為反應(yīng)-擴散過程,在這種過程中,少數(shù)的化學(xué)物質(zhì)會發(fā)生擴散并且相互反應(yīng);隨著簡單規(guī)則指導(dǎo)機體反應(yīng)的情況下,這些模型或許就能夠產(chǎn)生一些非常復(fù)雜的擬序結(jié)構(gòu),因此植物和動物的生物學(xué)結(jié)構(gòu)或許并不需要復(fù)雜的編程。
類似地,很顯然人類大腦中有著100萬億個連接,這才是真正制造我們的本源所在,但我們或許并不可能對其進行單一性地遺傳編程操作,近來一項在人工智能上的突破性研究就是基于大腦神經(jīng)網(wǎng)絡(luò)開展的,所謂人工智能就是擁有簡單元件(和神經(jīng)元類似)的大腦計算機模型,其能夠通過與外界相互作用來建立一定的聯(lián)系,而相關(guān)研究結(jié)果推測,在應(yīng)用領(lǐng)域內(nèi),比如手寫識別和醫(yī)療診斷中,Google公司就會邀請公眾來同人工智能計算機來打游戲以及觀察夢境。
微生物已經(jīng)超越了基礎(chǔ)的世界
因此,很顯然,單細胞并不需要非常復(fù)雜的數(shù)量來產(chǎn)生非常復(fù)雜的效應(yīng),那么人類機體基因的數(shù)量或許就和單細胞的微生物,比如病毒和細菌一樣,具有相同的尺寸。非常微小的微生物都有著極其豐富、復(fù)雜的生命,當(dāng)然近幾年不斷興起的社會微生物學(xué)就是研究微生物世界極其復(fù)雜“社會關(guān)系”的一門學(xué)科。
過去10年里研究者通過研究發(fā)現(xiàn),微生物會將其90%的生命以生物被膜的形式存在,這或許是一種最有效有力的一種生物學(xué)組織,的確許多生物被膜都同細胞間有著復(fù)雜的通訊機制,就像大腦組織一樣,這或許能夠啟發(fā)科學(xué)家們開發(fā)一種新型模型來研究諸如偏頭痛和癲癇癥等腦部障礙。
生物被膜被認為是“微生物的城市”,如今社會微生物以及相關(guān)的醫(yī)學(xué)研究近些年取得了快速的發(fā)展,比如囊性纖維化的療法開發(fā)商,居住在“城市”中的微生物也會互相協(xié)作、發(fā)生競爭、自殺等等一系列活動,因此其或許會成為21世紀科學(xué)家們在進化生物學(xué)中從事的重點領(lǐng)域研究之一。
人類的生物學(xué)機制或許遠比我們想象地要更加杰出,當(dāng)然對微生物世界的研究或許也會變得非常有趣,而關(guān)于基因的數(shù)量似乎同生命的奧秘并沒有什么關(guān)系。

編輯:趙彥
關(guān)鍵詞:基因 計數(shù) 人類




 中國制造助力孟加拉國首條河底隧道項目
中國制造助力孟加拉國首條河底隧道項目 澳大利亞豬肉產(chǎn)業(yè)協(xié)會官員看好進博會機遇
澳大利亞豬肉產(chǎn)業(yè)協(xié)會官員看好進博會機遇 聯(lián)合國官員說敘利亞約1170萬人需要人道主義援助
聯(lián)合國官員說敘利亞約1170萬人需要人道主義援助 伊朗外長扎里夫宣布辭職
伊朗外長扎里夫宣布辭職 中國南極中山站迎來建站30周年
中國南極中山站迎來建站30周年 聯(lián)合國特使赴也門斡旋荷臺達撤軍事宜
聯(lián)合國特使赴也門斡旋荷臺達撤軍事宜 以色列前能源部長因從事間諜活動被判11年監(jiān)禁
以色列前能源部長因從事間諜活動被判11年監(jiān)禁 故宮博物院建院94年來首開夜場舉辦“燈會”
故宮博物院建院94年來首開夜場舉辦“燈會”
 法蒂瑪·馬合木提
法蒂瑪·馬合木提 王召明
王召明 王霞
王霞 辜勝阻
辜勝阻 聶震寧
聶震寧 錢學(xué)明
錢學(xué)明 孟青錄
孟青錄 郭晉云
郭晉云 許進
許進 李健
李健 覺醒法師
覺醒法師 呂鳳鼎
呂鳳鼎 賀鏗
賀鏗 金曼
金曼 黃維義
黃維義 關(guān)牧村
關(guān)牧村 陳華
陳華 陳景秋
陳景秋 秦百蘭
秦百蘭 張自立
張自立 郭松海
郭松海 李蘭
李蘭 房興耀
房興耀 池慧
池慧 柳斌杰
柳斌杰 曹義孫
曹義孫 毛新宇
毛新宇 詹國樞
詹國樞 朱永新
朱永新 張曉梅
張曉梅 焦加良
焦加良 張連起
張連起 龍墨
龍墨 王名
王名 何水法
何水法 李延生
李延生 鞏漢林
鞏漢林 李勝素
李勝素 施杰
施杰 王亞非
王亞非 艾克拜爾·米吉提
艾克拜爾·米吉提 姚愛興
姚愛興 賈寶蘭
賈寶蘭 謝衛(wèi)
謝衛(wèi) 湯素蘭
湯素蘭 黃信陽
黃信陽 張其成
張其成 潘魯生
潘魯生 馮丹藜
馮丹藜 艾克拜爾·米吉提
艾克拜爾·米吉提 袁熙坤
袁熙坤 毛新宇
毛新宇 學(xué)誠法師
學(xué)誠法師 宗立成
宗立成 梁鳳儀
梁鳳儀 施 杰
施 杰 張曉梅
張曉梅


